2. EMP基本介绍
EasyMicroPlot致力于帮助低代码经验的微生物数据分析研究者,完成下游注释中一系列主流微生物常规分析流程。EMP包BUG提交及讨论QQ群:729506293
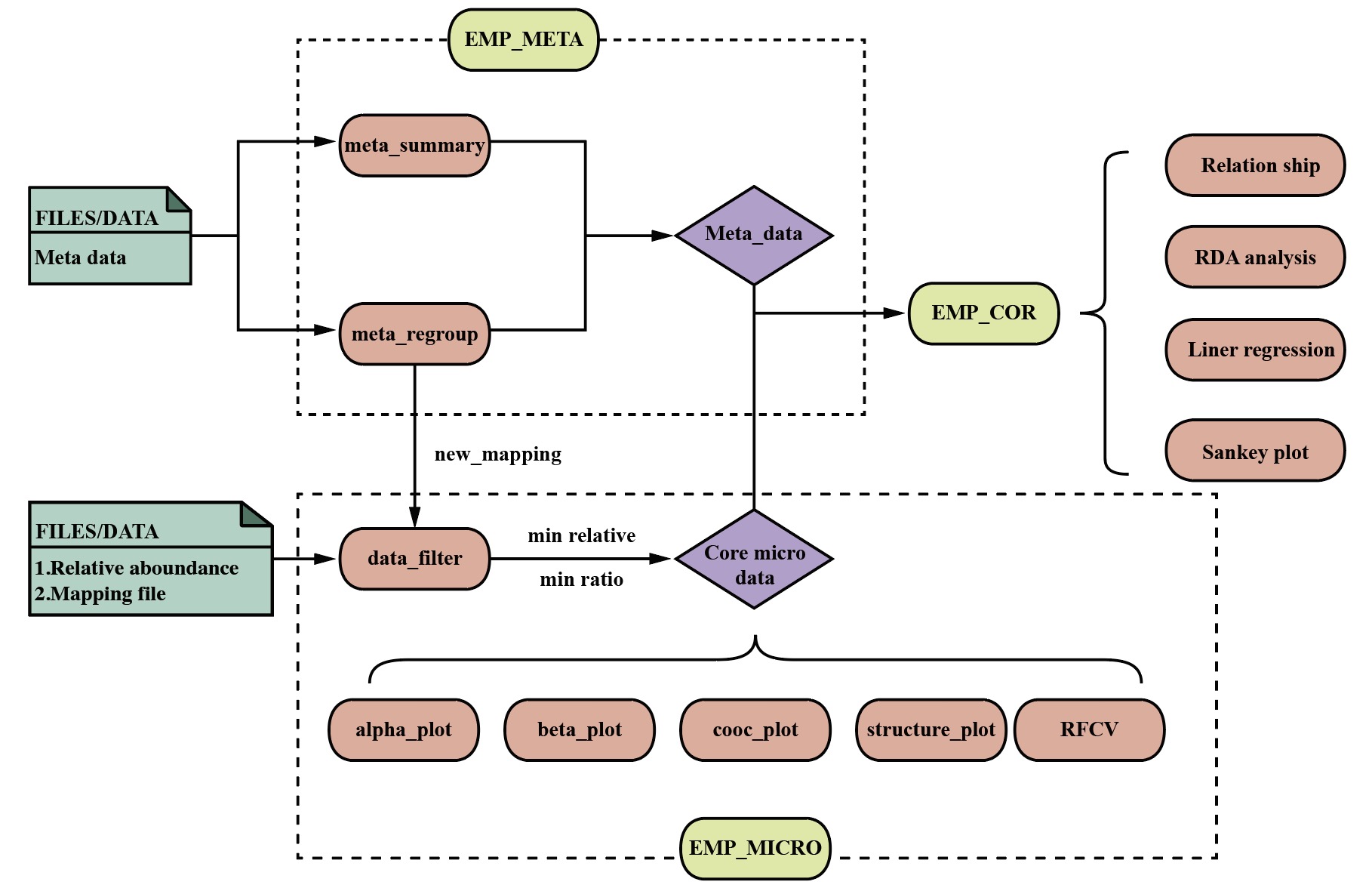
2.1 EMP主要架构
2.2 安装方式
Github安装方法
install.packages('remotes')
remotes::install_github("https://github.com/xielab2017/EasyMicroPlot",subdir='Version_0.5')
Gitee安装方法
考虑到github的网络连接问题,EMP包提供了中国境内Gitee安装方式
首先要确保电脑内已经安装Git工具
Git工具官方下载安装地址:https://git-scm.com/downloads (首次安装Git后,需重启电脑或者R)
install.packages('remotes')
remotes::install_git("https://gitee.com/xielab2017/EasyMicroPlot/",subdir='Version_0.5')
2.3 依赖包列表
- ggplot2
- vegan (>= 2.5-6)
- ape (>= 5.3)
- grid (>= 3.5.1)
- plyr (>= 1.8.6)
- dplyr (>= 1.0.2)
- multcomp (>= 1.4-14)
- patchwork (>= 1.0.1)
- fs (>= 1.5.0)
- stringr (>= 1.4.0)
- htmlwidgets (>= 1.5.3)
- ggiraph (>= 0.7.0)
- randomForest(>= 4.6-14)
- ggpubr (>= 0.4.0)
- purrr (>= 0.3.4)
- reshape2 (>= 1.4.4)
- psych (>= 2.0.12)
- VIM (>= 5.1.1)
- table1 (>= 1.4.2)
- pROC (>= 1.17.0.1)
- corrplot (>= 0.84)
- igraph (>= 1.2.6)
- filesstrings (>= 3.2.1)
- grid (>= 3.6.3)
- ggrepel (>= 0.9.1)
- RColorBrewer(>= 1.1-2)
- scales(>= 1.1.1)
- ggpmisc(>= 0.4.5)
- tidyr(>= 1.1.4)
- tibble(>= 3.1.6)
- networkD3(>= 0.4)
rlang(>= 1.0.6)
2.4 引用方式
Bingdong Liu, Liujing Huang, Zhihong Liu, Xiaohan Pan, Zongbing Cui, Jiyang Pan*,Liwei Xie*. EasyMicroPlot : An Efficient and Convenient R Package in Microbiome Downstream Analysis and Visualization for Clinical Study. Frontiers in Genetics. doi: 10.3389/fgene.2021.803627