13. 相关性分析图
微生物研究中,微生物数据常常和表型数据存在着广泛的线性关系。本章节EMP_COR模块提供两种展现微生物数据和表型数据相关性的方式。
13.1 EMP_COR模块
EMP_COR模块是可以根据4.1的data_filter函数筛选出各个级别的核心微生物并进行与表型数据构建相关性分析图。
13.1.1 参数介绍
data由
data_filter函数产生的包含各个样本微生物的数据框。meta样本的表型数据,格式需要符合
3.2格式要求。method相关性计算方式(pearson,spearman,kendall)。【默认:Spearman】
aes_value选择相关性热图或者三角形图,对应值为1和2。
width输出图形宽度。
height输出图形高度。
cor_output选择是否输出图形到工作区文件夹内。
file_name设定输出图形文件名。
13.1.2 使用范例
代码示例:
Tips1:由于表型数据样本和微生物数据样本往往不能完全一致,因此计算过程将采取二者样本的交集进行。
# 基本代码
library(EasyMicroPlot) # 加载包
# 加载表型数据
data(EMP)
meta_data <- EMP$iron # 这里使用内置的表型数据,用户也可以自行读取自己的数据,注意满足3.2格式要求
core_data <- data_filter(dir = '16s_data/',design = 'mapping/mapping.txt',
min_relative = 0.001,min_ratio = 0.7)
core_phylum <- core_data$filter_data$phylum # 这里用户可以自行选择所需要的微生物物种级别
# 两种不同相关性计算方案
## 微生物数据和表型数据分开进行关联分析 (热图形式)
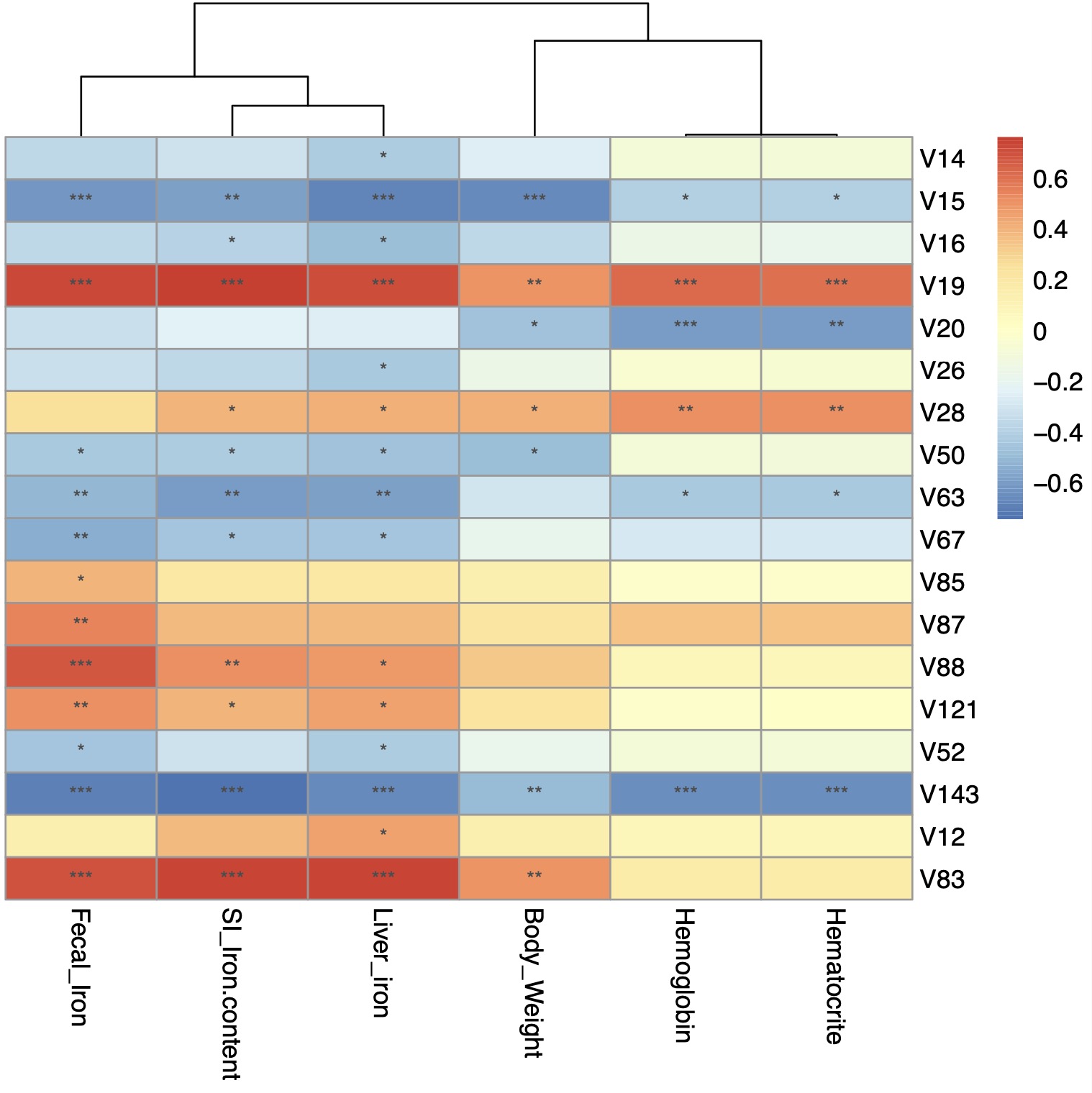
cor_re <- EMP_COR(data = core_phylum,meta=meta_data,cor_output = F,
method = 'spearman',aes_value = 1)
## 微生物数据和表型数据合并进行关联分析 (三角图形式)
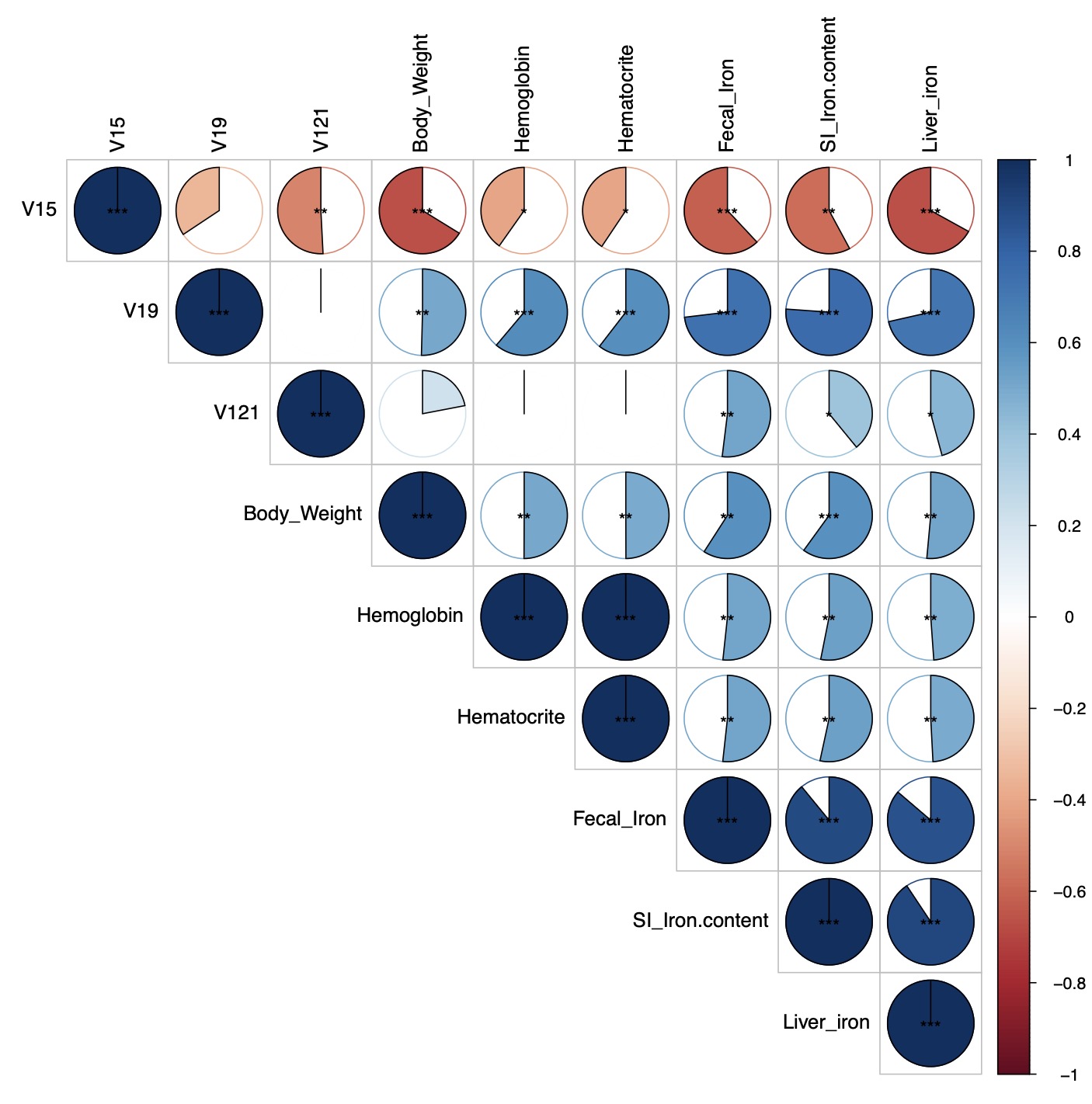
cor_re <- EMP_COR(data = core_phylum,meta=meta_data,cor_output = F,
method = 'spearman',aes_value = 2)
cor_re$r ## 相关性系数结果
cor_re$p ## 相关性检验结果
图形结果展示:
Tips 1: 这里的相关性图形仅保留通过相关性检验的结果,如1个物种或者表型数据与对面矩阵所有数据均无法通过相关性检验,则会被省略。详细结果可以在cor_re$p 查看。
# 生物数据和表型数据分开进行关联分析 (热图形式)
# 基本代码
library(EasyMicroPlot)
data(EMP)
meta_data <- EMP$iron # 这里使用内置的表型数据,用户也可以自行读取自己的数据,注意满足3.2格式要求
core_data <- data_filter(dir = '16s_data/',design = 'mapping/mapping.txt',
min_relative = 0.001,min_ratio = 0.7)
core_species <- core_data$filter_data$species # 这里用户可以自行选择所需要的微生物物种级别
cor_re <- EMP_COR(data = core_species,meta=meta_data,cor_output = F,
method = 'spearman',aes_value = 1)
# 生物数据和表型数据分开进行关联分析 (三角图形式)
# 基本代码
library(EasyMicroPlot)
data(EMP)
meta_data <- EMP$iron # 这里使用内置的表型数据,用户也可以自行读取自己的数据,注意满足3.2格式要求
core_data <- data_filter(dir = '16s_data/',design = 'mapping/mapping.txt',
min_relative = 0.001,min_ratio = 0.7)
core_species <- core_data$filter_data$species # 这里用户可以自行选择所需要的微生物物种级别
key_species <- subset(core_species,select = c('SampleID','V15','V19','V121')) # 也可以根据需要自定义需要的物种
cor_re <- EMP_COR(data = key_species,meta=meta_data,cor_output = F,
method = 'spearman',aes_value = 2)