14. 双变量拟合图
本章节提供EMP_COR_FIT模块提供可双变量线性拟合的模型及可视化结果。
14.1 EMP_COR_FIT模块
EMP_COR_FIT模块根据微生物数据和表型数据进行双变量拟合分析。
14.1.1 参数介绍
data由
data_filter函数产生的包含各个样本微生物的数据框。meta
样本表型数据,格式需要符合
3.2格式要求。formula
设定拟合方程的格式
formula = y~poly(x,1,raw = T)。eq_size
方程字体大小。
se
线性拟合是否添加置信区间。
height
图形高度。
width
图形宽度。
group
是否按照分组进行拟合。
palette
指定绘图色板。
14.1.2 使用范例
代码示例:
# 基本代码
library(EasyMicroPlot) # 加载包
# 加载表型数据
data(EMP)
meta_data <- EMP$iron # 这里使用内置的表型数据,用户也可以自行读取自己的数据,注意满足3.2格式要求
core_data <- data_filter(dir = '16s_data/',design = 'mapping/mapping.txt',
min_relative = 0.001,min_ratio = 0.7)
core_species <- core_data$filter_data$species # 这里用户可以自行选择所需要的微生物物种级别
# 一元一次拟合方程
fit_result <- EMP_COR_FIT(data=core_species,meta = meta_data,var_select = c('V19','SI_Iron.content'),
formula = y~poly(x,1,raw = T),width = 5,height = 5,se = F,group = F,eq_size = 3)
fit_result$pic # 基本拟合图形结果
fit_result$html # 交互式拟合图形结果
图形结果展示:
Tips 1: 下面的示例中,采用的是微生物的单个菌与表型数据的单个特征进行拟合,用户也可以采用微生物的两个菌之间进行拟合。
Tips 2: 当不需要输入表型数据时,可以在参数中设定meta = NULL。
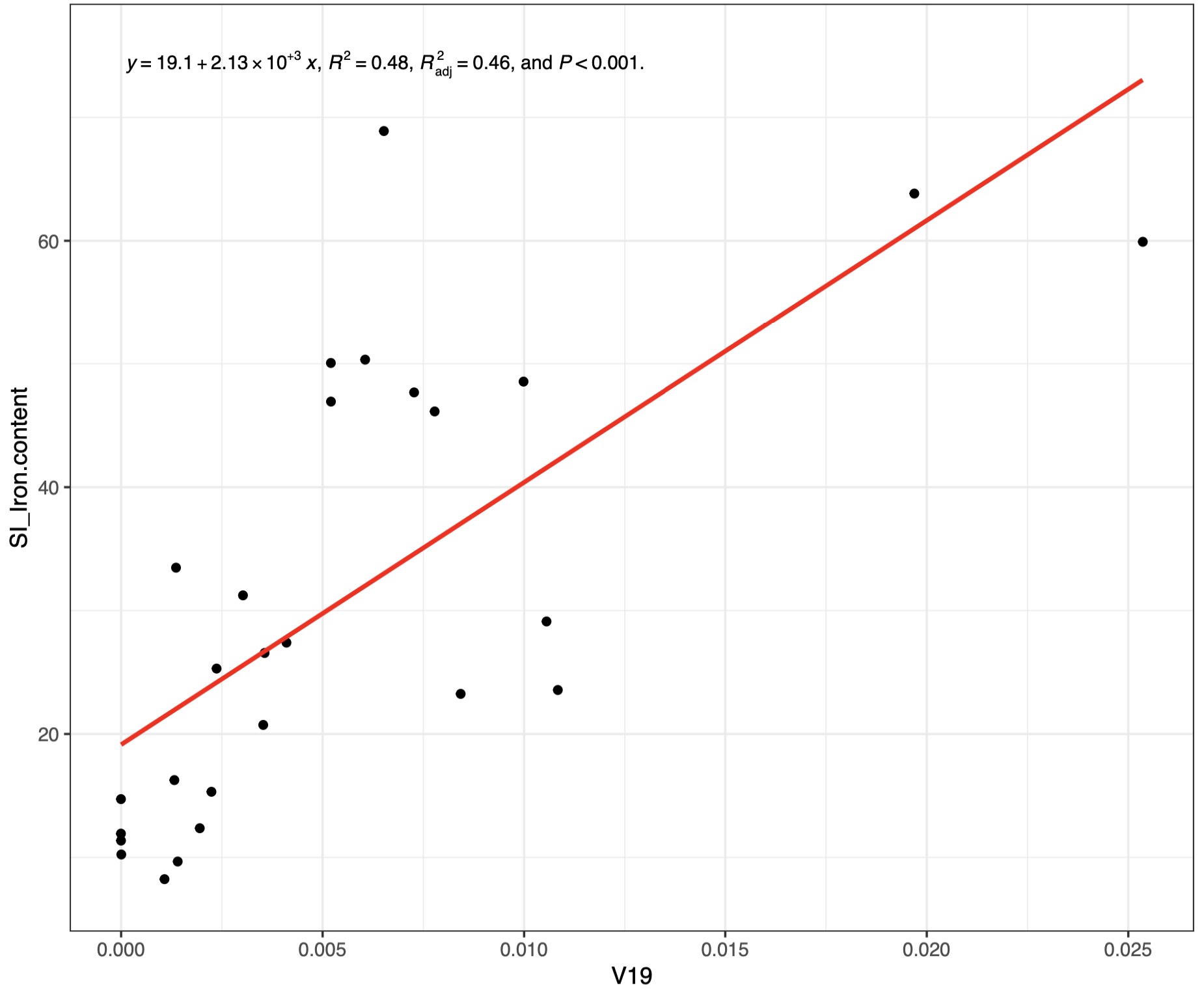
# 一元一次拟合方程
fit_result <- EMP_COR_FIT(data=core_species,meta = meta_data,var_select = c('V19','SI_Iron.content'),
formula = y~poly(x,1,raw = T),width = 5,height = 5,se = F,group = F,eq_size = 3)
fit_result$pic
# 交互式图形结果
fit_result$html
Tips 3:可以将鼠标放在感兴趣的点,查询样本信息及基本情况。
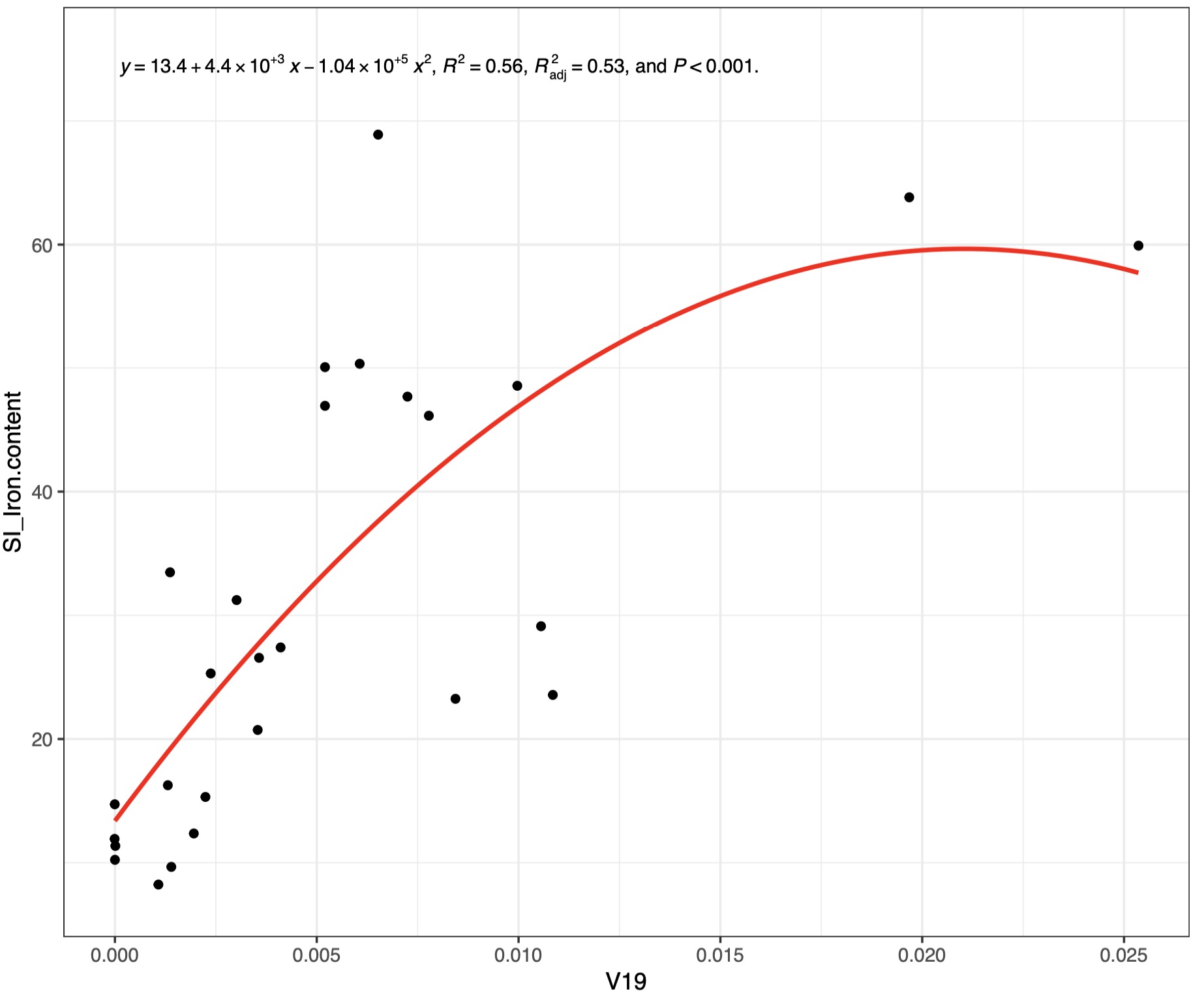
# 一元二次拟合方程
fit_result <- EMP_COR_FIT(data=core_species,meta = meta_data,var_select = c('V19','SI_Iron.content'),
formula = y~poly(x,2,raw = T),width = 5,height = 5,se = F,group = F,eq_size = 3)
fit_result$pic
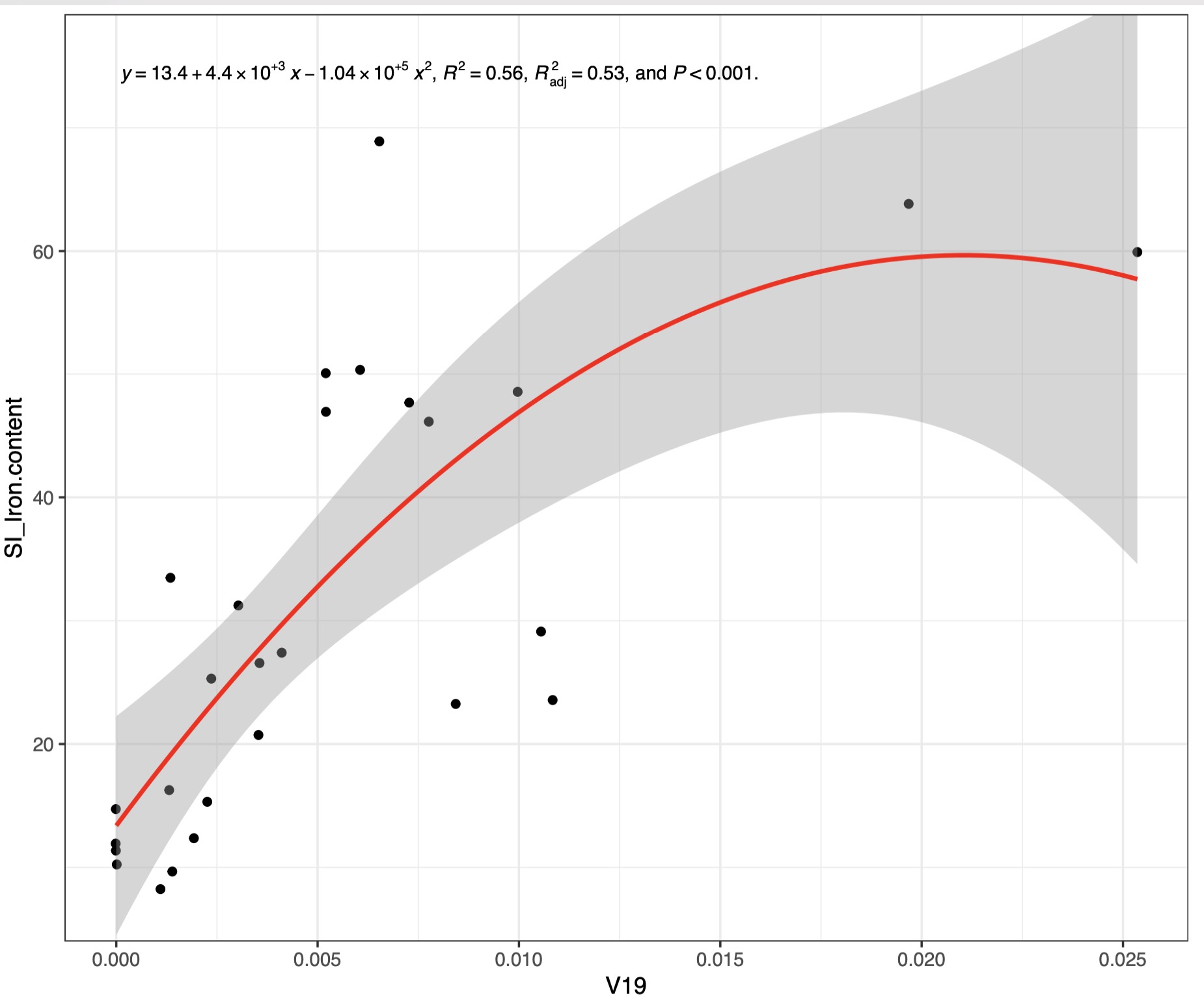
# 增加置信区间
fit_result <- EMP_COR_FIT(data=core_species,meta = meta_data,var_select = c('V19','SI_Iron.content'),
formula = y~poly(x,2,raw = T),width = 5,height = 5,se = T,group = F,eq_size = 3)
fit_result$pic
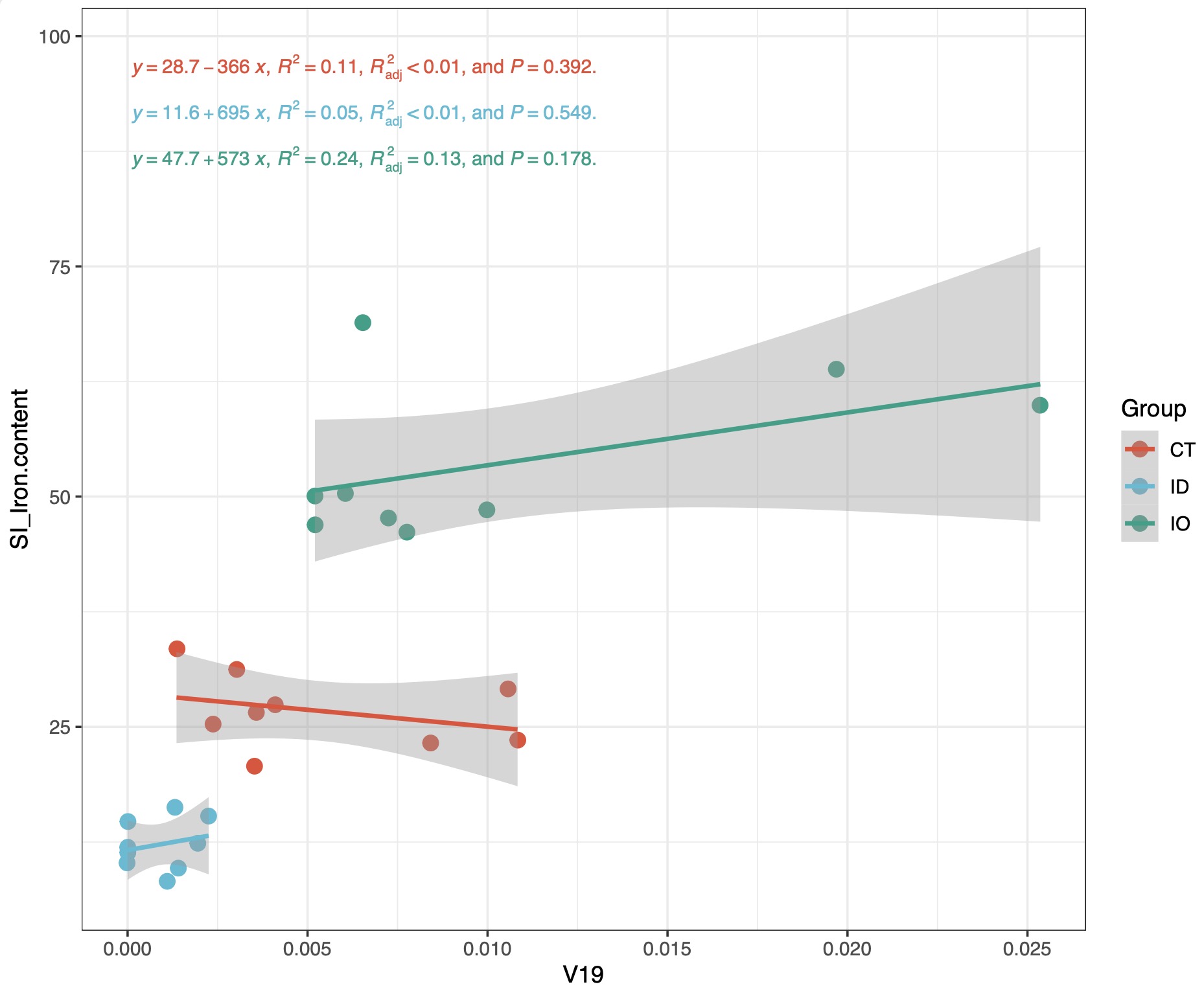
# 采用分组数据拟合模型
fit_result <- EMP_COR_FIT(data=core_species,meta = meta_data,var_select = c('V19','SI_Iron.content'),
formula = y~poly(x,1,raw = T),width = 5,height = 5,se = T,group = T,eq_size = 3)
fit_result$pic