8. Beta多样性
β多样性常用来评估组间微生物结构的差异性。本章节beta_plot模块提供了多种距离算法用于进行维度差异统计及可视化结果。
8.1 beta_plot模块
beta_plot模块是基于4.1的data_filter函数进行beta多样性计算,即可衔接data_filter函数计算结果,也可直接仿照data_filter函数语法,读取dataframe、list和file三种格式。
8.1.1 参数介绍
Tips 1: beta_plot模块提供了过程数据结果,因此用户可以手动计算后,自行标注统计结果。
data输入微生物数据支持
dataframe和list两种格式,dataframe应符合3.1中的格式要求,也可将不同级别微生物数据dataframe直接合并为list输入。dir:指定微生物数据文件存放的地址,文件可以以
csv和txt格式保存,格式应符合3.1中的格式要求。min_relative:指定最小的微生物物种相对丰度数值,低于此阈值的相对丰度将会被过滤为0。
min_ratio:指定最小的微生物物种在至少一个分组内的出现率,低于此阈值的物种将会被过滤。
design:指定样本分组信息
mapping文件,支持直接输入dataframe或者指定文件地址。ellipse:设置散点的置信区间(0~1)。【默认:Null】
adjust:在
min_relative和min_ratio过滤下,部分样品的微生物物种可能过滤为空,打开此选项可以将空样本赋予一个特殊特征在beta多样性中以区分彼此。【默认:True】method:此参数将决定多样性差异统计的方法,(ttest,HSD, LSD,duncan,scheffe,REGW,SNK)
distance:指定beta多样性计算过程中采用的距离运算。(bray,manhattan, euclidean, canberra, clark, kulczynski, jaccard, gower, altGower, morisita, horn, mountford, raup, binomial, chao, cao or mahalanobis)
pattern:指定在文件夹内微生物数据文件名的特征信息,与
dir配合使用output:指定是否将核心微生物计算结果输出。【默认:关闭】
html_out:指定是在工作区域文件夹内输出html结果。【默认:关闭】
width:指定html图形的宽度。
height:指定html图形的高度。
change:Qiime2产生物种注释结果中有时出现完全为空的情况(
d__Bacteria;__;__),打开此选项后可以将其修正为(d__Bacteria;Other;Other)。【默认:关闭】change_name:指定空注释的修改名,与
change配合使用。【默认:Other】row_panel:指定拼图结果输出每行的图形数目。
seed:指定随机数种子,便于确定图形中散点的随机分布。【默认:123】
group_level:指定图形中分组显示的排列顺序。
palette:指定绘图色板。
8.1.2 使用范例
Tips 2:以下均在种级别进行演示,实际使用用户可以自行查看其他级别结果。代码示例:
library(EasyMicroPlot) # 加载包
beta_re <- beta_plot(dir = '16s_data/',design = 'mapping/mapping.txt',
min_relative = 0.001,min_ratio = 0.7,
distance = 'bray',method = 'LSD')
beta_re$result$filter_data ## 这里存储了前置data_filter函数过滤的核心微生物结果
beta_re$plot ## 这里存储了核心微生物各个级别beta多样性的图形结果
基本计算结果:
# 种级别各个样本核心微生物beta多样性的前三个维度的坐标结果
beta_re$plot$species$pc_data
sample PC1 PC2 PC3 Group
A01 A01 0.23325291 0.035594497 0.030648810 CT
A02 A02 -0.09285486 -0.103393814 -0.031886190 CT
A03 A03 0.04045387 -0.125091827 -0.085172668 CT
A04 A04 0.05209699 0.002954595 -0.031074589 CT
A05 A05 -0.17985725 -0.042200037 -0.093639030 CT
A06 A06 0.14258227 -0.012533821 -0.051640448 CT
A07 A07 0.17424665 -0.081945583 -0.112918281 CT
A08 A08 0.11542759 0.077427298 0.046016115 CT
A09 A09 0.26532418 -0.058154301 0.119570046 CT
B10 B10 -0.22348190 -0.095896824 -0.033245259 ID
B11 B11 -0.03644198 0.290161251 -0.039450952 ID
B12 B12 -0.10341177 0.241732219 0.038140553 ID
B13 B13 0.03959259 -0.055961831 -0.126781260 ID
B14 B14 -0.07445329 0.061620012 0.128671704 ID
B15 B15 -0.02873585 0.133737593 -0.007086424 ID
B16 B16 -0.04350455 0.305562428 -0.052682899 ID
B17 B17 -0.23365667 0.027446065 0.120774234 ID
B18 B18 -0.12871343 0.220700157 -0.011274409 ID
C19 C19 0.09098629 -0.011571871 -0.025207411 IO
C20 C20 0.25263686 0.057607262 -0.021869906 IO
C21 C21 0.08242074 -0.104046547 0.067203835 IO
C22 C22 0.12718637 -0.034803390 0.024611743 IO
C23 C23 0.13368309 -0.116743577 0.124434349 IO
C24 C24 -0.12367366 -0.156461584 -0.024632571 IO
C25 C25 -0.17801061 -0.185940261 0.031275942 IO
C26 C26 -0.23549301 -0.114908182 0.069901074 IO
C27 C27 -0.06760157 -0.154889926 -0.052686107 IO
# 种级别各个样本核心微生物beta多样性的三个轴事后检验的详细结果
# 第一轴
beta_re$plot$species$test$PC1_test
difference pvalue signif. LCL UCL
CT - ID 0.17594213 0.0123 * 0.04174051 0.31014375
CT - IO 0.07428198 0.2646 -0.05991964 0.20848360
ID - IO -0.10166015 0.1310 -0.23586177 0.03254147
# 第二轴
beta_re$plot$species$test$PC2_test
difference pvalue signif. LCL UCL
CT - ID -0.15960490 0.0036 ** -0.2615115 -0.05769826
CT - IO 0.05715723 0.2584 -0.0447494 0.15906386
ID - IO 0.21676213 0.0002 *** 0.1148555 0.31866876
# 第三轴
beta_re$plot$species$test$PC3_test
difference pvalue signif. LCL UCL
CT - ID -0.02524017 0.4688 -0.09600133 0.04552099
CT - IO -0.04479191 0.2038 -0.11555307 0.02596925
ID - IO -0.01955174 0.5738 -0.09031290 0.05120942
Tips 3: 这里提取前三个维度的坐标结果也可以进行其他统计计算,例如T检验或者Kruskal-Wallis检验。
# Kruskal-Wallis Rank Sum Test
location_data <-beta_re$plot$species$pc_data
kruskal.test(PC1~Group,location_data) # 第一轴的计算
Kruskal-Wallis rank sum test
data: PC1 by Group
Kruskal-Wallis chi-squared = 5.9718, df = 2, p-value = 0.05049
# T test
location_data <-beta_re$plot$species$pc_data
t.test(PC1~Group,location_data[1:18,]) # CT组和ID组在第一轴的计算
Welch Two Sample t-test
data: PC1 by Group
t = 3.0639, df = 13.356, p-value = 0.008813
alternative hypothesis: true difference in means between group CT and group ID is not equal to 0
95 percent confidence interval:
0.05222005 0.29966421
sample estimates:
mean in group CT mean in group ID
0.08340804 -0.09253409
图形结果展示:
# 种级别beta多样性结果
beta_re <- beta_plot(dir = '16s_data/',design = 'mapping/mapping.txt',
min_relative = 0.001,min_ratio = 0.7,
distance = 'bray',method = 'LSD')
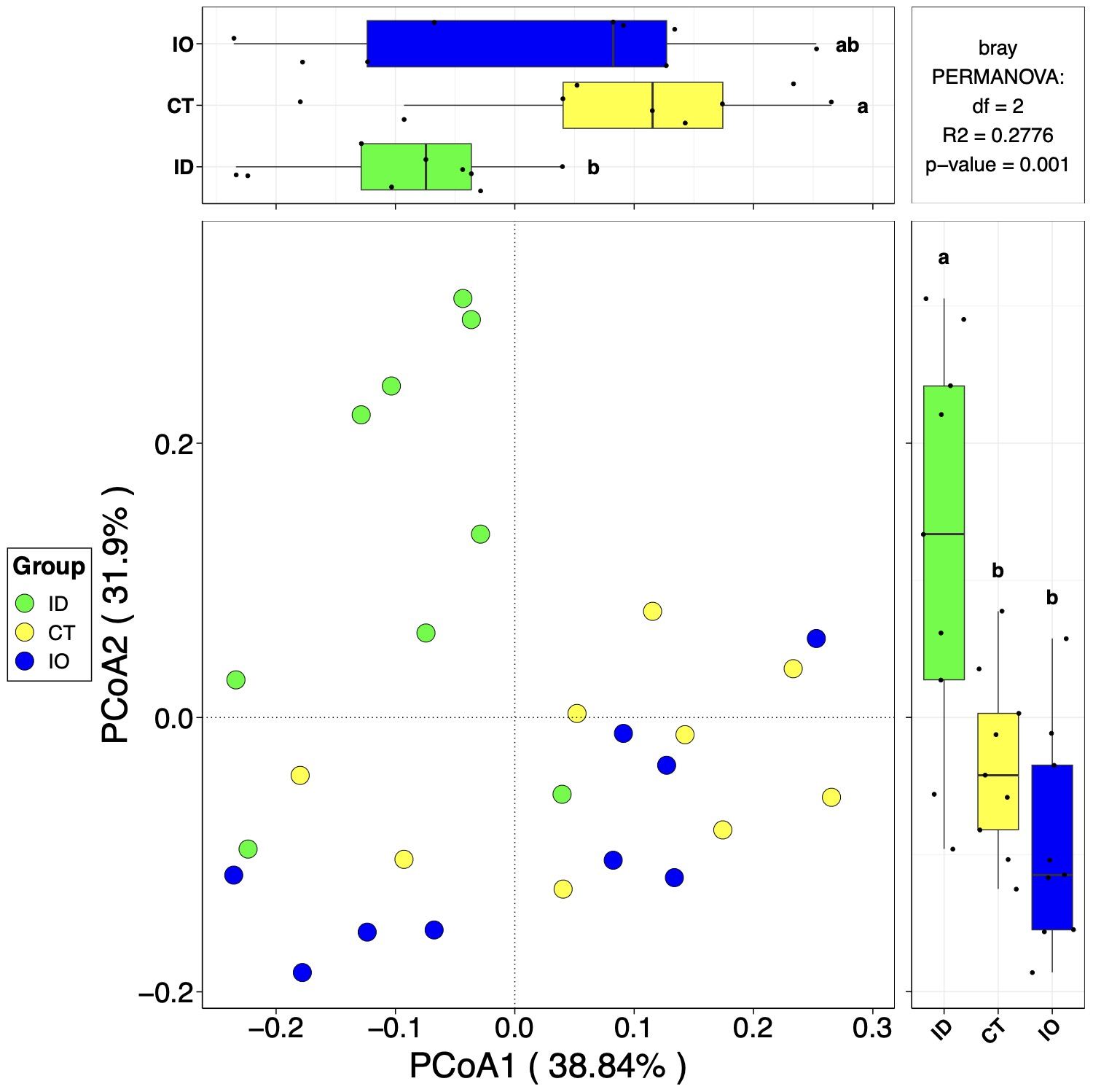
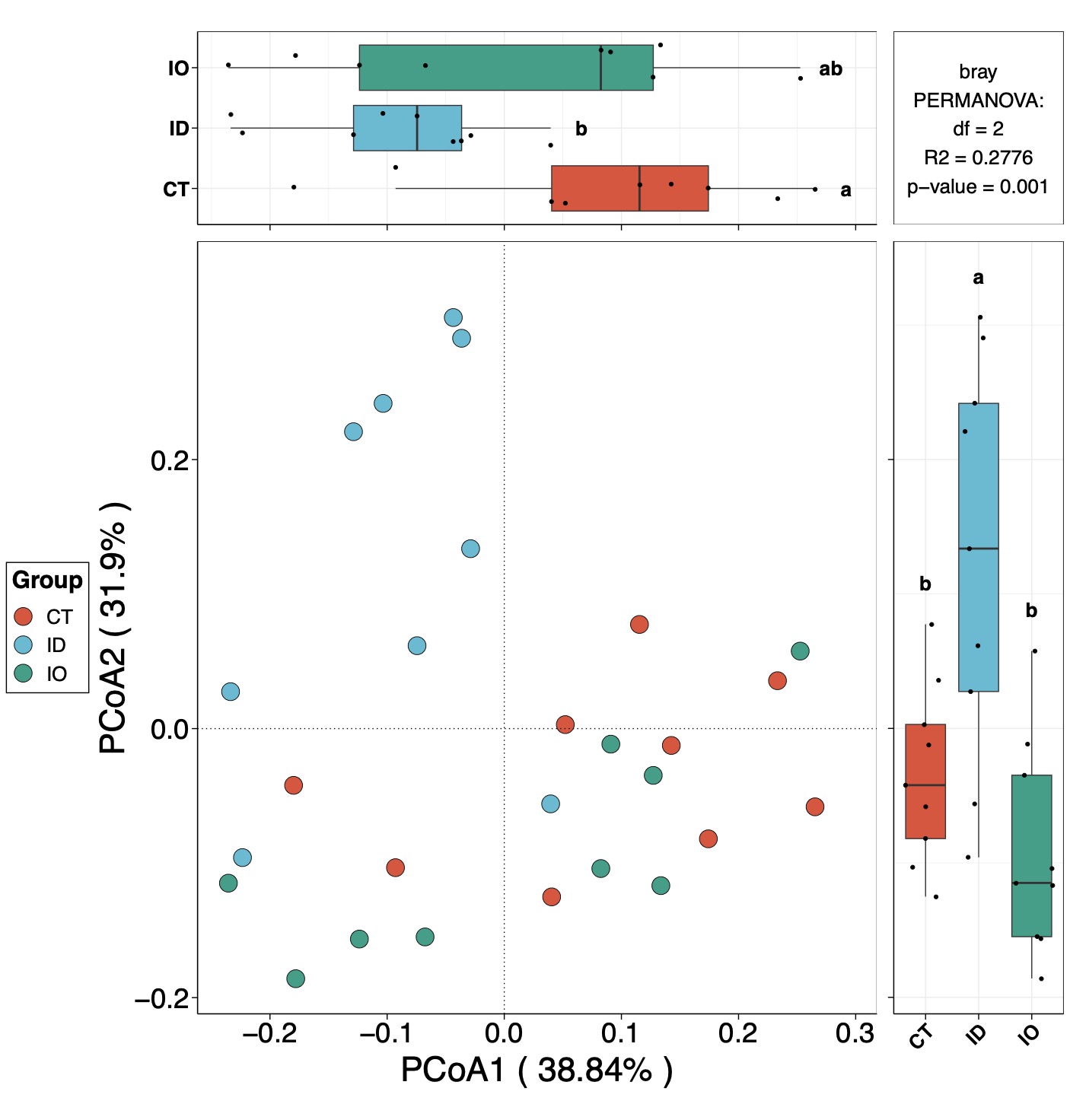
beta_re$plot$species$pic #这里储存了三个轴所组成的三个平面的图形结果
# 最常用的第一和第二轴组成的平面结果,用户也可以自行查看其他两个平面结果
# 图形右上角为Adonis分析的结果
beta_re$plot$species$pic$p12 # 由于Rstudio窗口的限制,用户需要调整窗体以达到最优的美化结果
# 或者直接输出合适尺寸的pdf结果,在工作文件夹内查看
ggplot2::ggsave(beta_re$plot$species$pic$p12,
filename = 'beta_p12.pdf',width = 15,height = 15)
# 结果也支持输出交互式图形,便于发现每个样本的具体情况
beta_re$plot$species$html$p12
Tips 4:可以将鼠标放在感兴趣的点,查询样本信息及基本情况。
# 图形输出可以通过调整group_level参数和palette参数进行调整分组排序和颜色方案
group_order <- c('ID','CT','IO') ## 设定排序,名称需要绝对一致
cols <- c('green','yellow','blue') ## 设定颜色方案
beta_re <- beta_plot(dir = '16s_data/',design = 'mapping/mapping.txt',
min_relative = 0.001,min_ratio = 0.7,
distance = 'bray',method = 'LSD',
group_level = group_order,palette = cols)
# 建议直接输出合适尺寸的pdf结果,在工作文件夹内查看
ggplot2::ggsave(beta_re$plot$species$pic$p12,filename = 'beta_p12.pdf',width = 15,height = 15)
# 或者直接查看交互式html格式
beta_re$plot$species$pic$p12