9. Co-oc共发生网络
共发生网络分析是利用相关性检验评估微生物物种之间的竞争或者抑制关系,也可以利用图论相关技术来从整体评估微生物网络的基本属性,筛选可能存在的微生物社区网络及网络关键微生物。
9.1 cooc_plot模块
cooc_plot模块是基于4.1的data_filter函数筛选出各个级别的核心微生物,再基于相关系数并用igraph包进行beta多样性计算,因此可以直接仿照data_filter函数语法,读取dataframe、list和file三种格式。
9.1.1 参数介绍
data输入微生物数据支持
dataframe和list两种格式,dataframe应符合3.1中的格式要求,也可将不同级别微生物数据dataframe直接合并为list输入。meta样本的表型数据,格式需要符合
3.2格式要求。dir:指定微生物数据文件存放的地址,文件可以以
csv和txt格式保存,格式应符合3.1中的格式要求。min_relative:指定最小的微生物物种相对丰度数值,低于此阈值的相对丰度将会被过滤为0。
min_ratio:指定最小的微生物物种在至少一个分组内的出现率,低于此阈值的物种将会被过滤。
design:指定样本分组信息
mapping文件,支持直接输入dataframe或者指定文件地址。group_combie:打开这个参数可以将全部分组合并为一个数据集进行共发生网络计算。【默认:False】
clust:打开这个参数可以共发生网络的节点根据
fast greedy算法进行聚类。【默认:False】cooc_method:指定节点之间相关性计算方式(person,spearman,kendall)。【默认:Spearman】
cooc_p:设定节点之间相关性计算的p值阈值,高于此阈值的相关性结果将会被过滤。【默认:0.05】
cooc_r:设定节点之间相关性计算的相关系数绝对值的阈值,低于此阈值的相关性结果将会被过滤。【默认:0.3】
vertex.size:设定共发生网络中节点的大小。
vertex.label.cex:设定共发生网络中节点注释文字大小。
edge.width:设定共发生网络中边的宽度。【默认:2】
edge.curved:设定边是否为曲线。【默认:False】
seed:设定随机数种子,便于确定图形中散点的随机分布。【默认:123】
width,height:设定共发生网络图的宽和高。【默认:10】
heatmap_width,heatmap_height:设定共发生网络图的节点重要性评估热图的宽和高。
set_color_level:设定共发生网络中节点按照微生物注释级别进行染色。【默认:phylum】
edge_color_positive设定正相关边的颜色。
edge_color_negetive设定负相关边的颜色。
meta_col:设定表型数据在共发生网络中的节点颜色。【默认:white】
adjust:在
min_relative和min_ratio过滤下,部分样品的微生物物种可能过滤为空,打开此选项可以将空样本赋予一个特殊特征在beta多样性中以区分彼此。【默认:False】pattern:指定在文件夹内微生物数据文件名的特征信息,与
dir配合使用output:指定是否将核心微生物计算结果输出。【默认:False】
cooc_output:指定共发生网络分析的结果输出文件夹名字。【默认:cooc_result】
change:Qiime2产生物种注释结果中有时出现完全为空的情况(
d__Bacteria;__;__),打开此选项后可以将其修正为(d__Bacteria;Other;Other)。【默认:False】change_name:指定空注释的修改名,与
change配合使用。【默认:Other】
9.1.2 使用范例
代码示例:
library(EasyMicroPlot) # 加载包
cooc_re <- cooc_plot(dir = '16s_data/',design = 'mapping/mapping.txt',
min_relative = 0.001,min_ratio = 0.7,
cooc_method = 'spearman',cooc_output = T,
cooc_p = 0.05,cooc_r = 0.3)
cooc_re$result$filter_data ## 这里存储了前置data_filter函数过滤的核心微生物结果
cooc_re$cooc_profile ## 这里存储了核心微生物各个级别共发生网络的基本属性结果
cooc_re$plot ## 这里存储了核心微生物各个级别共发生网络的igraph相关图形信息
基本计算结果:
Tips 1: 这里仅展示种级别CT组的结果,用户可以根据输入数据情况查看其他级别的结果。
Tips 2: 当group_combie = True时,所有组的数据将合并处理,组名将统一为Total
# 种级别各个分组的网络基本属性
cooc_re$cooc_profile$species
num.vertices num.edges transitivity centralization_degree graph_density
CT 31 95 0.6756757 0.1956989 0.2043011
ID 26 57 0.4912892 0.2246154 0.1753846
IO 32 112 0.6211973 0.2903226 0.2258065
# 种级别CT组共发生网络节点之间相关性系数结果
cor_r <- cooc_re$plot$CT$species$cor_result$cor_r
# 种级别CT组共发生网络节点之间相关性系数统计结果
cor_p <- cooc_re$plot$CT$species$cor_result$cor_p
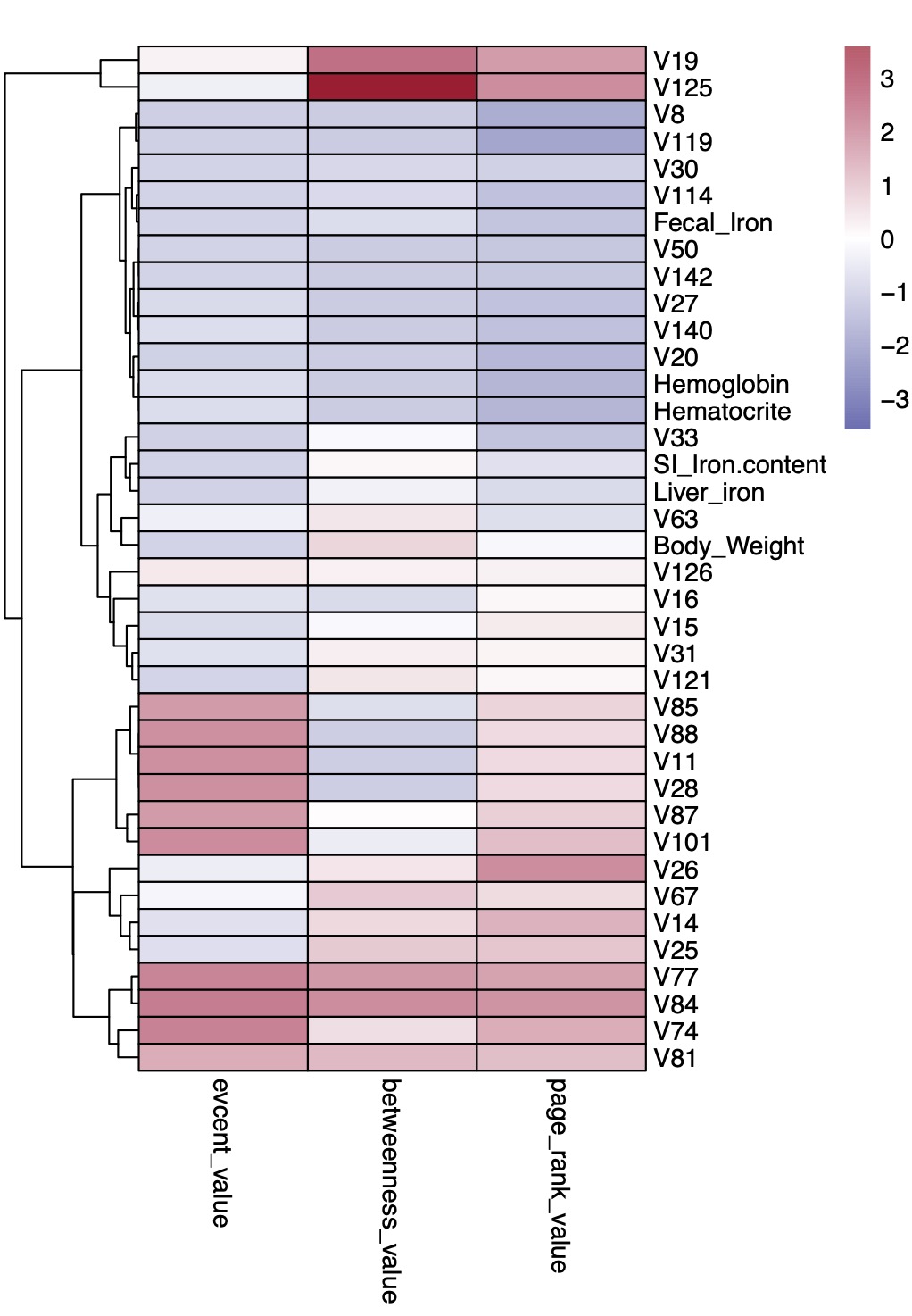
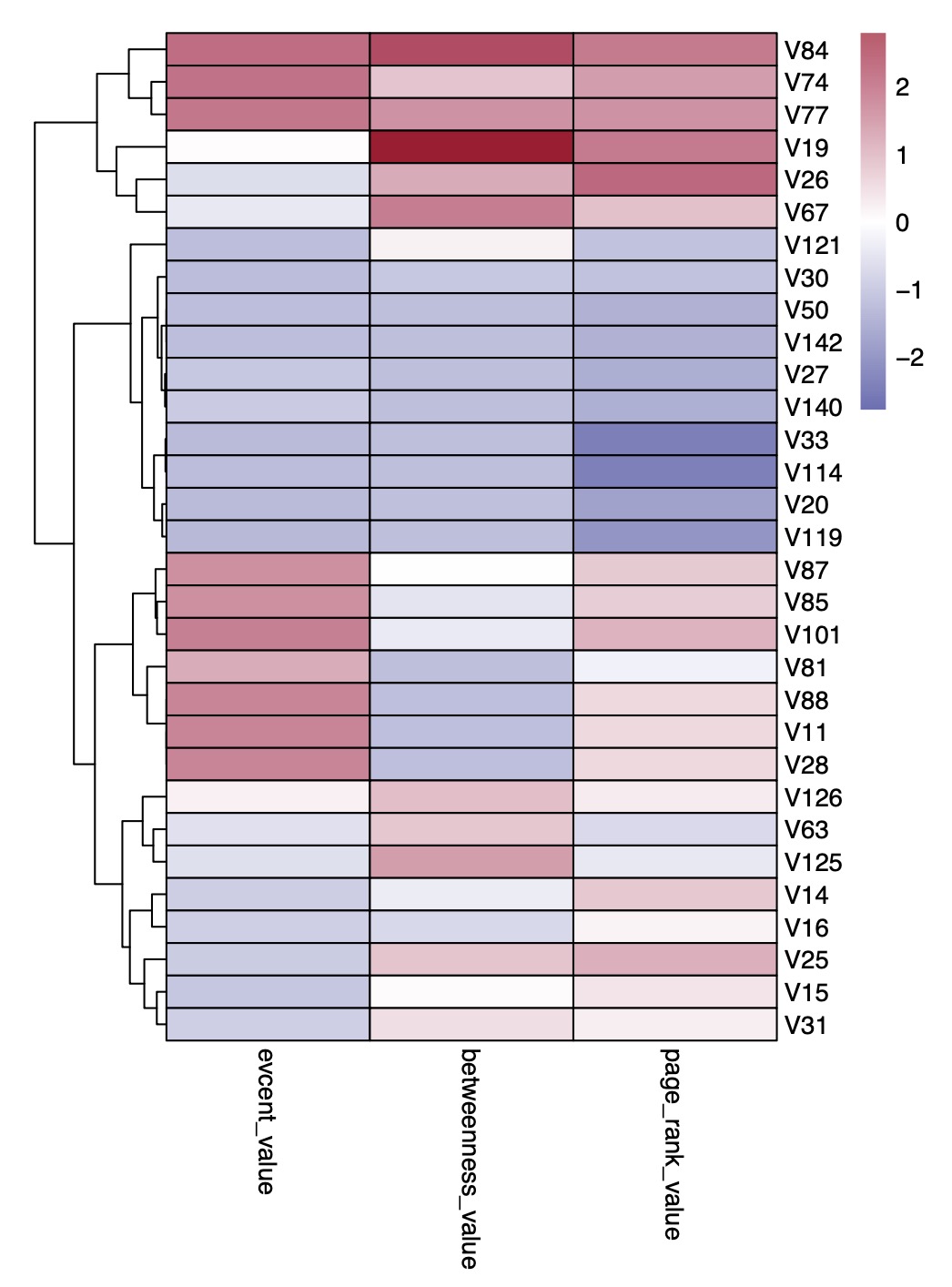
# 种级别CT组共发生网络中各个节点在不同算法的重要性评估
# EMP包内置了evcent_value,betweenness_value和page_rank_value三个常用的网络节点评估算法
cooc_re$plot$CT$species$vertex_attribute$vertex_importance_value
evcent_value betweenness_value page_rank_value
V11 0.894767363 0.0006568144 0.03748984
V14 0.111946208 0.0408464535 0.03996182
V15 0.073028000 0.0598905309 0.03606126
V16 0.120083019 0.0226038346 0.03390773
V19 0.375088867 0.2397686661 0.05081571
V20 0.010881847 0.0007662835 0.01612598
V25 0.101058494 0.1005016259 0.04333433
V26 0.194527714 0.1187298368 0.05377930
V27 0.076653472 0.0000000000 0.01853288
V28 0.894767363 0.0006568144 0.03748984
V30 0.024882835 0.0073508484 0.02180678
V31 0.118581669 0.0812700827 0.03444455
V33 0.011231679 0.0000000000 0.01010074
V50 0.025741613 0.0000000000 0.01887667
V63 0.211304738 0.0983519270 0.02584497
V67 0.249218353 0.1530166779 0.04089875
V74 0.975072258 0.1000790431 0.04602047
V77 0.958146550 0.1370721047 0.04759485
V81 0.726127159 0.0000000000 0.02994727
V84 1.000000000 0.1911416981 0.05093498
V85 0.843272919 0.0332010045 0.03918532
V87 0.842729507 0.0572413793 0.03966348
V88 0.894767363 0.0006568144 0.03748984
V101 0.915902846 0.0394804244 0.04287798
V114 0.023484488 0.0000000000 0.01033077
V119 0.003116891 0.0000000000 0.01409915
V121 0.028044633 0.0666666667 0.02178926
V125 0.205871904 0.1287356322 0.02830766
V126 0.427769779 0.1052228823 0.03480820
V140 0.096860597 0.0000000000 0.01860294
V142 0.025741613 0.0000000000 0.01887667
图形结果展示:
Tips 3: 由于EMP包的共发生网络主要基于igragh包进行计算,因此图形将会直接生成在工作区域的本地目录中,后续的版本更新计划中会添加对于ggplot2的支持。
# 各级别分组的共发生网络图形结果将会在工作区域的本地目录中生成
library(EasyMicroPlot)
cooc_re <- cooc_plot(dir = '16s_data/',design = 'mapping/mapping.txt',
min_relative = 0.001,min_ratio = 0.7,
cooc_method = 'spearman',cooc_output = T,clust = F,
cooc_p = 0.05,cooc_r = 0.3)
cooc_result/ # 共发生网络结果文件夹
├── network # 包含全部分组各个微生物级别核心微生物的共发生网络分析图
└── vertex # 包含全部分组各个微生物级别核心微生物的三种不同节点重要性评估结果热图
Tips 4: 为了图形美化,微生物节点用编码显示,可以在cooc_re$result$filter_data内查询具体微生物注释信息。例如种级别物种注释信息为cooc_re$result$filter_data$species_ID
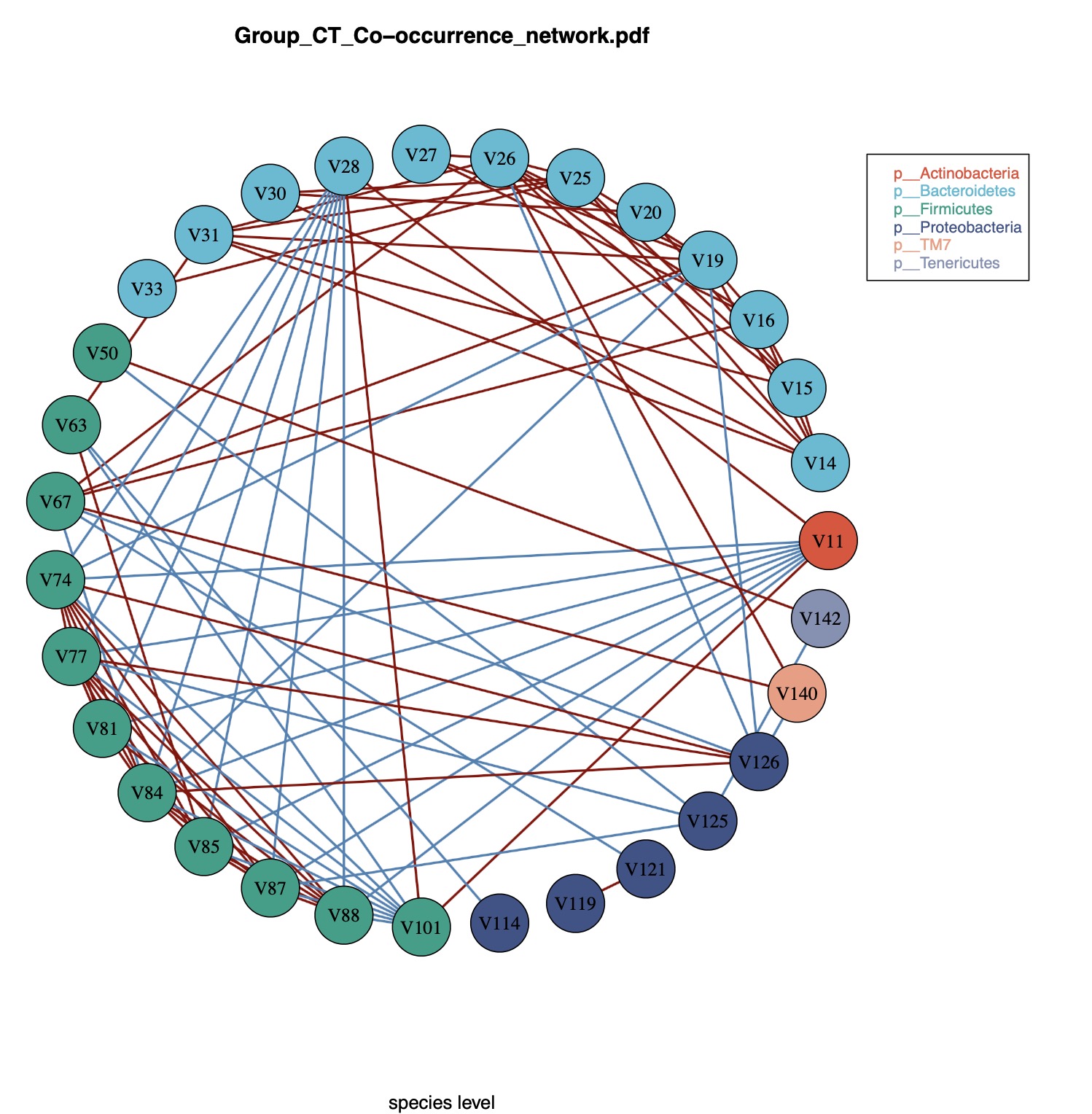
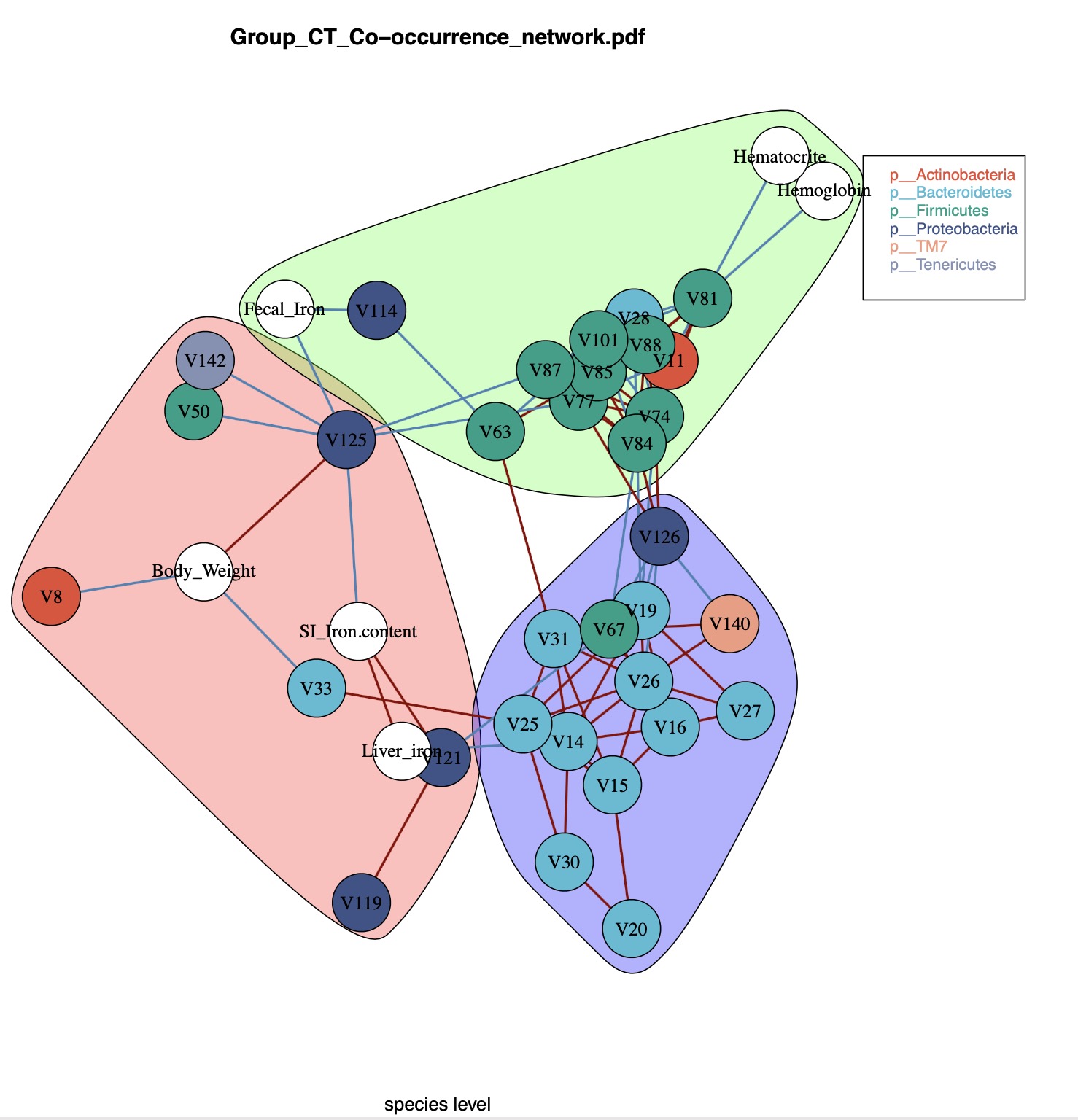
Tips 5: EMP包默认采用圆形排布展示共发生网络分析结果,当clust = True时候图形将根据fast greedy算法进行聚类。
# 注意修改为clust = T
library(EasyMicroPlot)
cooc_re <- cooc_plot(dir = '16s_data/',design = 'mapping/mapping.txt',
min_relative = 0.001,min_ratio = 0.7,cooc_method = 'spearman',
cooc_output = T,clust = T,
cooc_p = 0.05,cooc_r = 0.3)
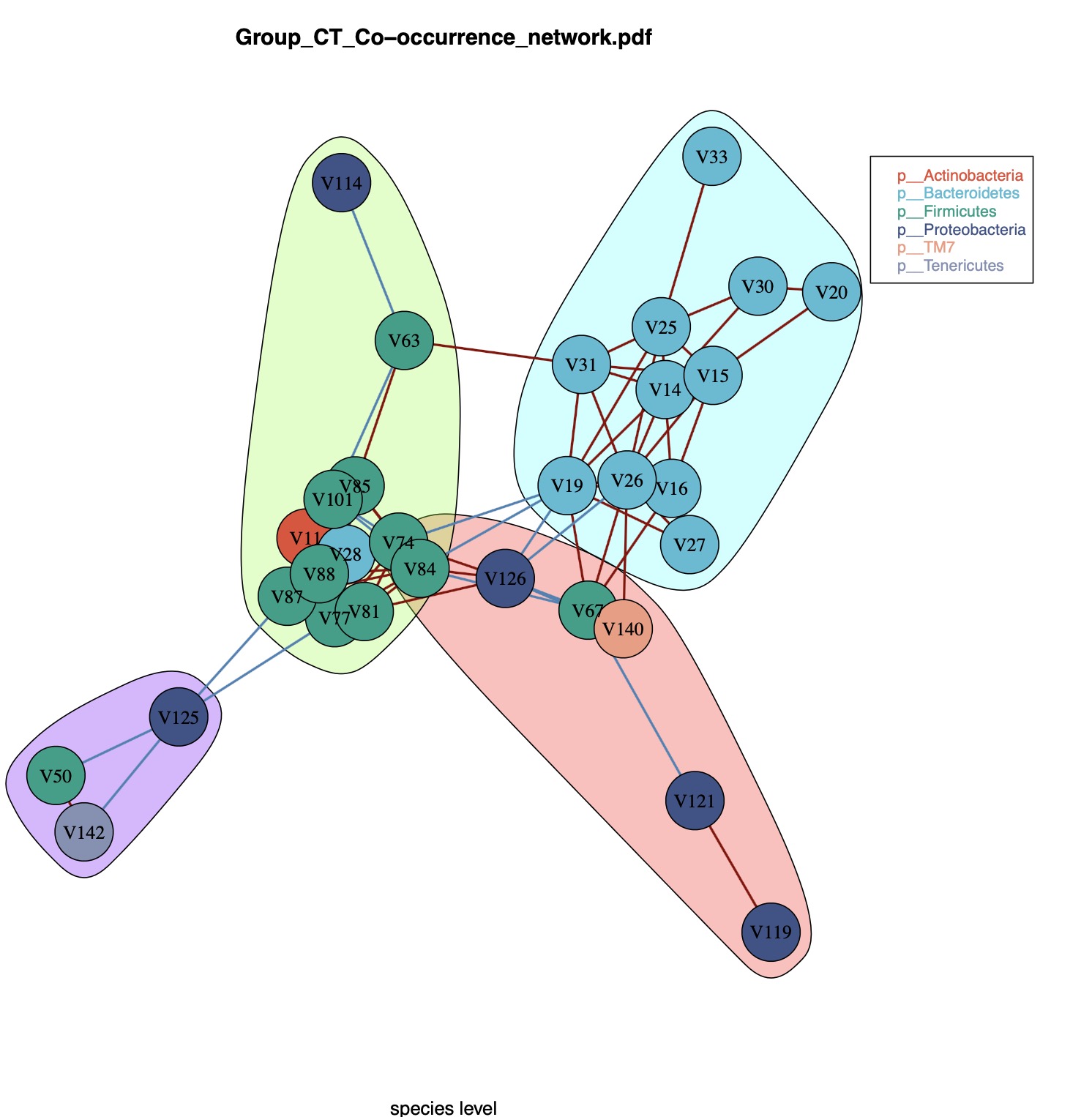

Tips 5: EMP包默认采用圆形排布展示共发生网络分析结果,当clust = True时候图形将根据fast greedy算法进行聚类。
# 为了方便直接查看图形结果和美化,EMP也内置cooc_plot_each快速对于特定的结果进行图形调整
# 例如可以直接对于CT组种级别图形进行部分参数的重新设定
cooc_plot_each(cooc_re$plot$CT$species,cooc_output=F,vertex.size = 12,
vertex.label.cex =1 ,edge.width =3,clust = T,
edge.curved = T,edge_color_positive = '#C34A36',
edge_color_negitive ='#845EC2')
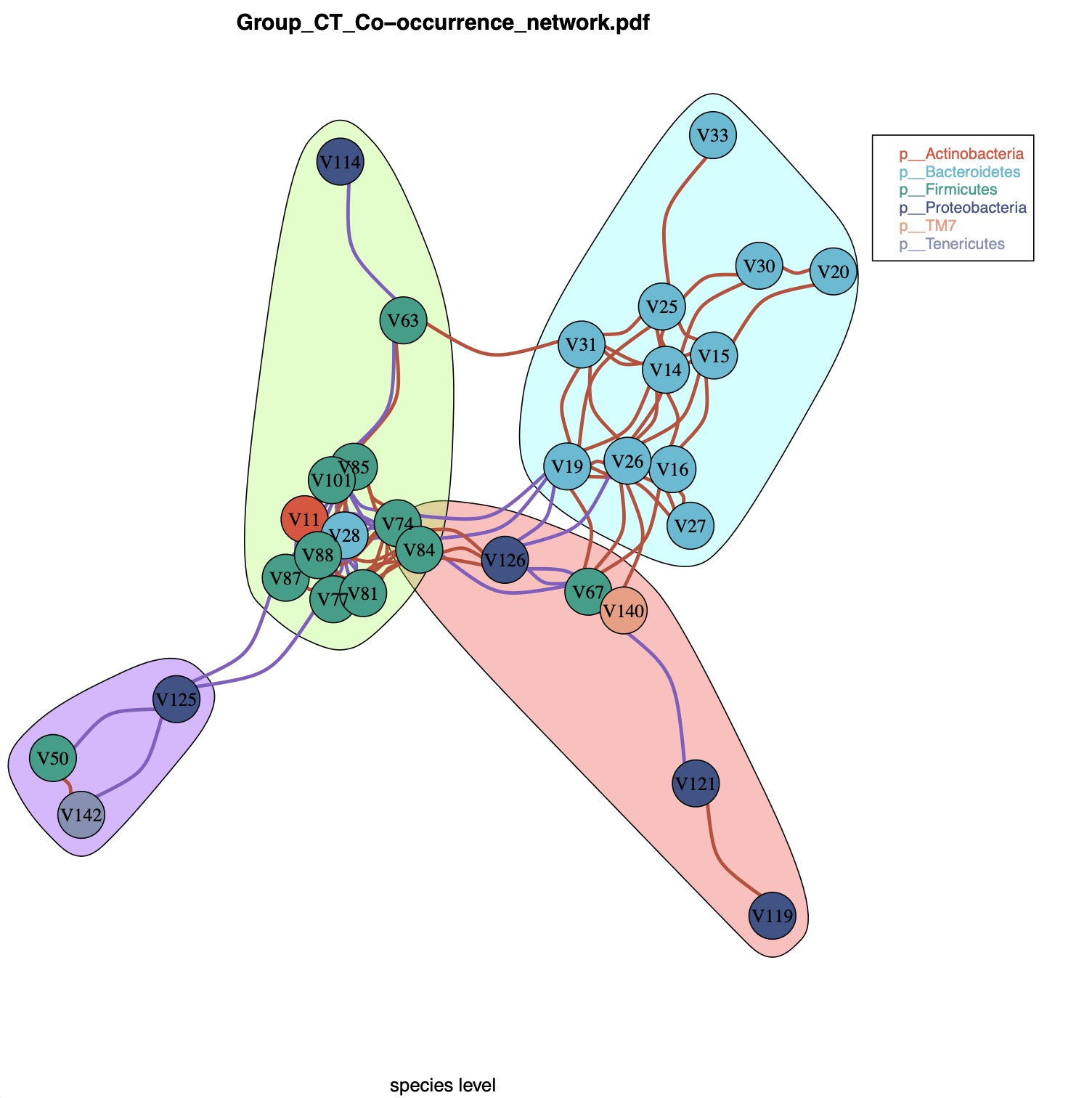
Tips 6: EMP包支持将表型数据与微生物数据联合在一起做共发生网络分析。由于表型数据样本和微生物数据样本往往不能完全一致,因此计算过程将采取二者样本的交集进行。
# 整合表型数据联合分析
meta_data <- EMP$iron # 注意要符合3.2的格式要求
cooc_re <- cooc_plot(dir = '16s_data/',design = 'mapping/mapping.txt',meta = meta_data,
min_relative = 0.001,min_ratio = 0.7,cooc_method = 'spearman',cooc_output = T,
cooc_p = 0.05,cooc_r = 0.3,clust = T )