7. Alpha多样性
α多样性是微生物下游分析中常见的分析方法,主要用于评估物种的丰富度。本章节alpha_plot模块基于相对丰度数据,内置了4种常见的α多样性的方法,并提供了常规的统计分析和可视化功能。
7.1 alpha_plot模块
alpha_plot模块是基于4.1的data_filter函数进行alpha多样性计算,即可衔接data_filter函数计算结果,也可直接仿照data_filter函数语法,读取dataframe、list和file三种格式。
7.1.1 参数介绍
data输入微生物数据支持
dataframe和list两种格式,dataframe应符合3.1中的格式要求,也可将不同级别微生物数据dataframe直接合并为list输入。dir:指定微生物数据文件存放的地址,文件可以以
csv和txt格式保存,格式应符合3.1中的格式要求。min_relative:指定最小的微生物物种相对丰度数值,低于此阈值的相对丰度将会被过滤为0。
min_ratio:指定最小的微生物物种在至少一个分组内的出现率,低于此阈值的物种将会被过滤。
design:指定样本分组信息
mapping文件,支持直接输入dataframe或者指定文件地址。adjust:在
min_relative和min_ratio过滤下,部分样品的微生物物种可能过滤为空,打开此选项可以将空样本赋予一个特殊特征在beta多样性中以区分彼此。【默认:False】method:此参数将决定多样性差异统计的方法,主要包含T检验(ttest)和onewayANOVA及多重检验(HSD, LSD,duncan,scheffe,REGW,SNK)
pattern:指定在文件夹内微生物数据文件名的特征信息,与
dir配合使用output:指定是否将核心微生物计算结果输出。【默认:False】
html_out:指定是在结果中输出html结果。【默认:False】
change:Qiime2产生物种注释结果中有时出现完全为空的情况(
d__Bacteria;__;__),打开此选项后可以将其修正为(d__Bacteria;Other;Other)。【默认:False】change_name:指定空注释的修改名,与
change配合使用。【默认:Other】row_panel:指定拼图结果输出每行的图形数目。
seed:指定随机数种子,便于确定图形中散点的随机分布。【默认:123】
group_level:指定图形中分组显示的排列顺序。
palette:指定绘图色板。
mytheme:支持ggplot2主题代码,便于图形美化。
7.1.2 使用范例
代码示例:
Tips 1:alpha_plot模块与data_filter模块一样支持dataframe、list和file三种格式。
Tips 2:以下均在种级别进行演示,实际使用用户可以自行查看其他级别结果。
Tips 3:method参数可以指定成LSD等方差分析的统计方式。
library(EasyMicroPlot) # 加载包
alpha_re <- alpha_plot(dir = '16s_data/',design = 'mapping/mapping.txt',min_relative = 0.001,min_ratio = 0.7,
method = 'ttest')
alpha_re$result$filter_data ## 这里存储了前置data_filter函数过滤的核心微生物结果
alpha_re$result$alpha_result ## 这里存储了核心微生物各个级别alpha多样性的计算结果
alpha_re$plot ## 这里存储了核心微生物各个级别alpha多样性的图形结果
基本计算结果:
# 种级别各个样本核心微生物alpha多样性结果
alpha_re$result$alpha_result$species
Pielou Shannon Simpson InvSimpson
A01 0.6948243 2.429458 0.8176936 7.485272
A02 0.7426636 2.596729 0.8862084 8.787994
A03 0.7076148 2.452406 0.8545843 6.876838
A04 0.7631435 2.691119 0.8724602 7.840688
A05 0.7837826 2.740502 0.9059144 10.628623
A06 0.7209308 2.520740 0.8454894 6.472048
A07 0.6536974 2.285658 0.8120621 7.320907
A08 0.6371104 2.227661 0.7835866 4.620785
A09 0.5946119 2.096816 0.7714934 4.376242
B10 0.7829667 2.713556 0.9069157 10.742948
B11 0.6321257 2.190781 0.7945742 4.867937
B12 0.6694254 2.360635 0.8143036 7.385135
B13 0.7716491 2.649833 0.8867659 8.831258
B14 0.6920953 2.440578 0.8603441 7.160455
B15 0.7316281 2.601193 0.8699751 7.690835
B16 0.6226102 2.138035 0.7871505 4.698154
B17 0.7231726 2.506325 0.8731743 7.884838
B18 0.6677607 2.354765 0.8126828 7.338539
C19 0.7366446 2.619028 0.8544939 6.872564
C20 0.6591650 2.263564 0.7867330 4.688959
C21 0.6828007 2.407802 0.8427408 6.358927
C22 0.6930151 2.483433 0.8253161 7.724627
C23 0.6610389 2.368845 0.8191883 7.530616
C24 0.7519618 2.651688 0.8923070 9.285650
C25 0.7335167 2.564747 0.8895906 9.057199
C26 0.7492045 2.641965 0.8972072 9.728307
C27 0.7194006 2.557720 0.8769097 8.124116
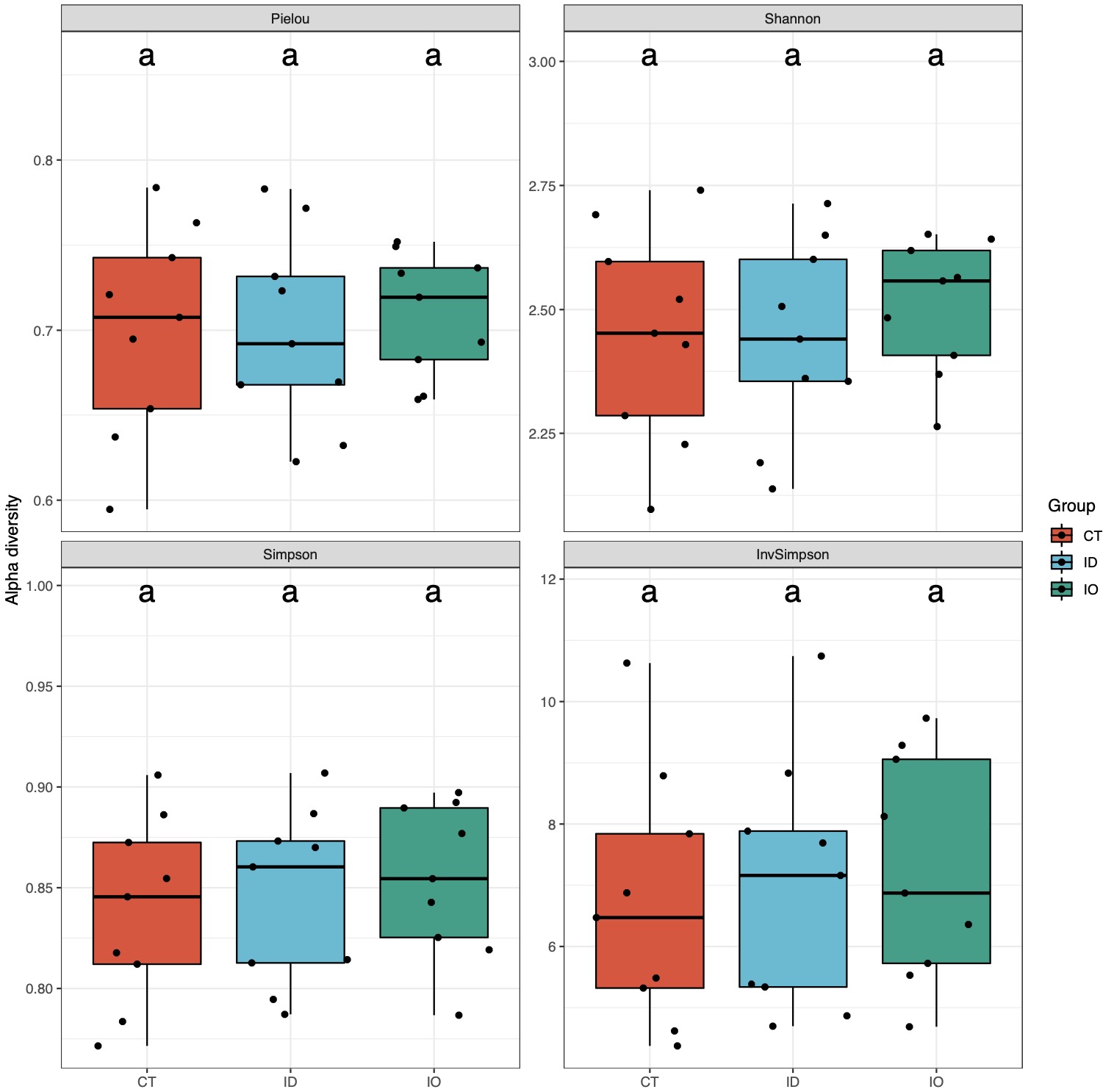
基本统计结果(仅适用于方差分析):
# 函数提供了各个微生物级别的onewayANOVA的详细记录
alpha_re$plot$species$test$ANOVA
$Pielou
Df Sum Sq Mean Sq F value Pr(>F)
Group 2 0.00061 0.0003063 0.109 0.897
Residuals 24 0.06740 0.0028081
$Shannon
Df Sum Sq Mean Sq F value Pr(>F)
Group 2 0.0237 0.01183 0.341 0.715
Residuals 24 0.8331 0.03471
$Simpson
Df Sum Sq Mean Sq F value Pr(>F)
Group 2 0.00102 0.0005108 0.28 0.758
Residuals 24 0.04384 0.0018265
$InvSimpson
Df Sum Sq Mean Sq F value Pr(>F)
Group 2 1.37 0.687 0.173 0.842
Residuals 24 97.21 3.967
# 函数提供了各个微生物级别的事后检验的详细记录
alpha_re$plot$species$test$comparison
$Pielou
difference pvalue signif. LCL UCL
CT - ID 0.0005494902 0.9826 -0.05100785 0.05210683
CT - IO -0.0098187401 0.6977 -0.06137608 0.04173860
ID - IO -0.0103682303 0.6818 -0.06192557 0.04118911
$Shannon
difference pvalue signif. LCL UCL
CT - ID 0.009487593 0.9149 -0.1717834 0.1907586
CT - IO -0.057522482 0.5187 -0.2387935 0.1237486
ID - IO -0.067010075 0.4529 -0.2482811 0.1142610
$Simpson
difference pvalue signif. LCL UCL
CT - ID -0.006265957 0.7585 -0.04784704 0.03531512
CT - IO -0.014999340 0.4638 -0.05658042 0.02658174
ID - IO -0.008733383 0.6685 -0.05031446 0.03284770
$InvSimpson
difference pvalue signif. LCL UCL
CT - ID -0.2434114 0.7977 -2.181290 1.694467
CT - IO -0.5512853 0.5626 -2.489164 1.386593
ID - IO -0.3078740 0.7458 -2.245752 1.630004
图形结果展示:
## 种级别alpha多样性结果
alpha_re <- alpha_plot(dir = '16s_data/',design = 'mapping/mapping.txt',
min_relative = 0.001,min_ratio = 0.7,
method = 'ttest')
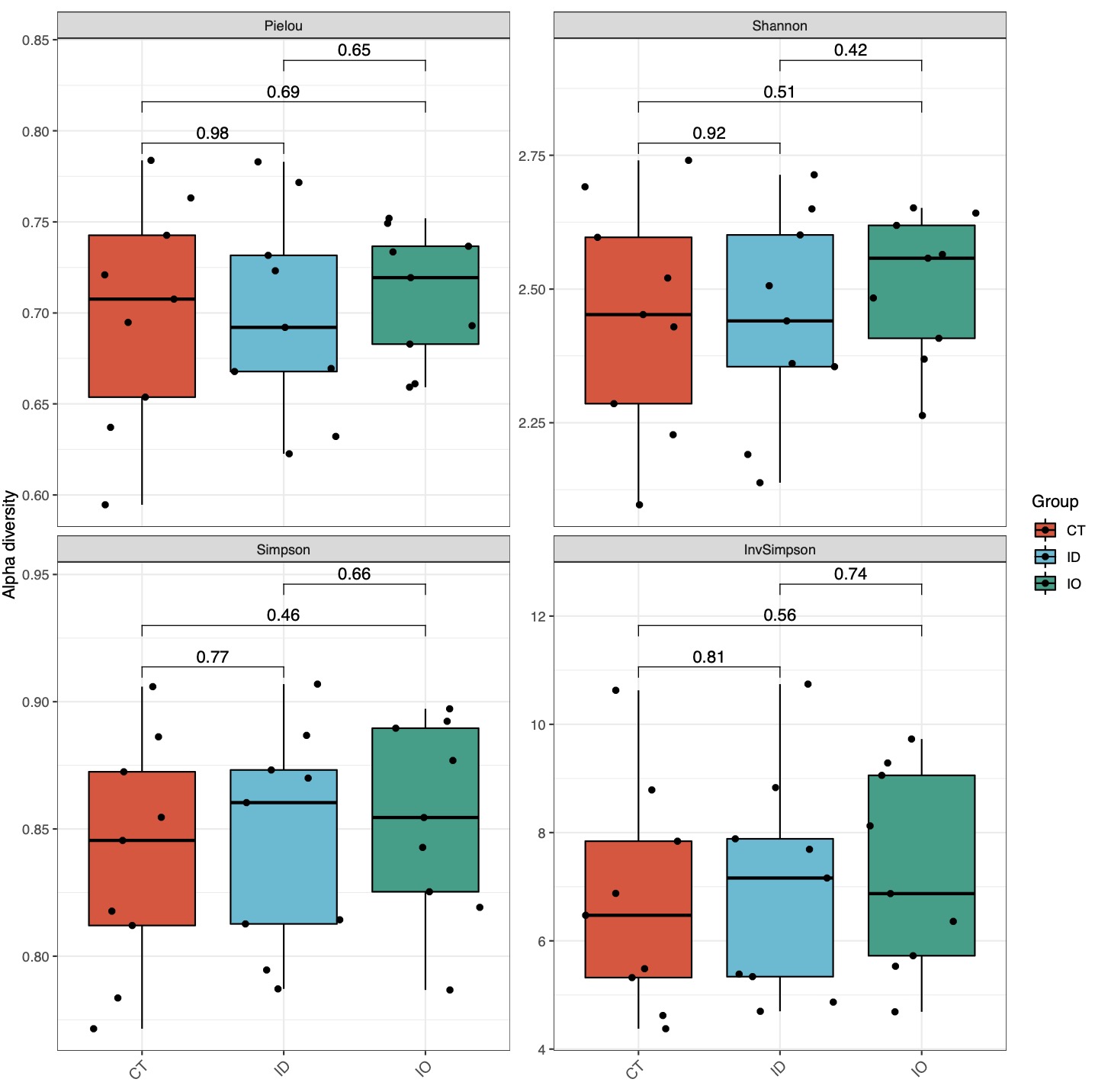
alpha_re$plot$species$pic$Total
# 结果也支持输出交互式图形,便于发现离群样本等情况
alpha_re$plot$species$html$Total
Tips 4:可以将鼠标放在感兴趣的点,查询样本信息及基本情况。
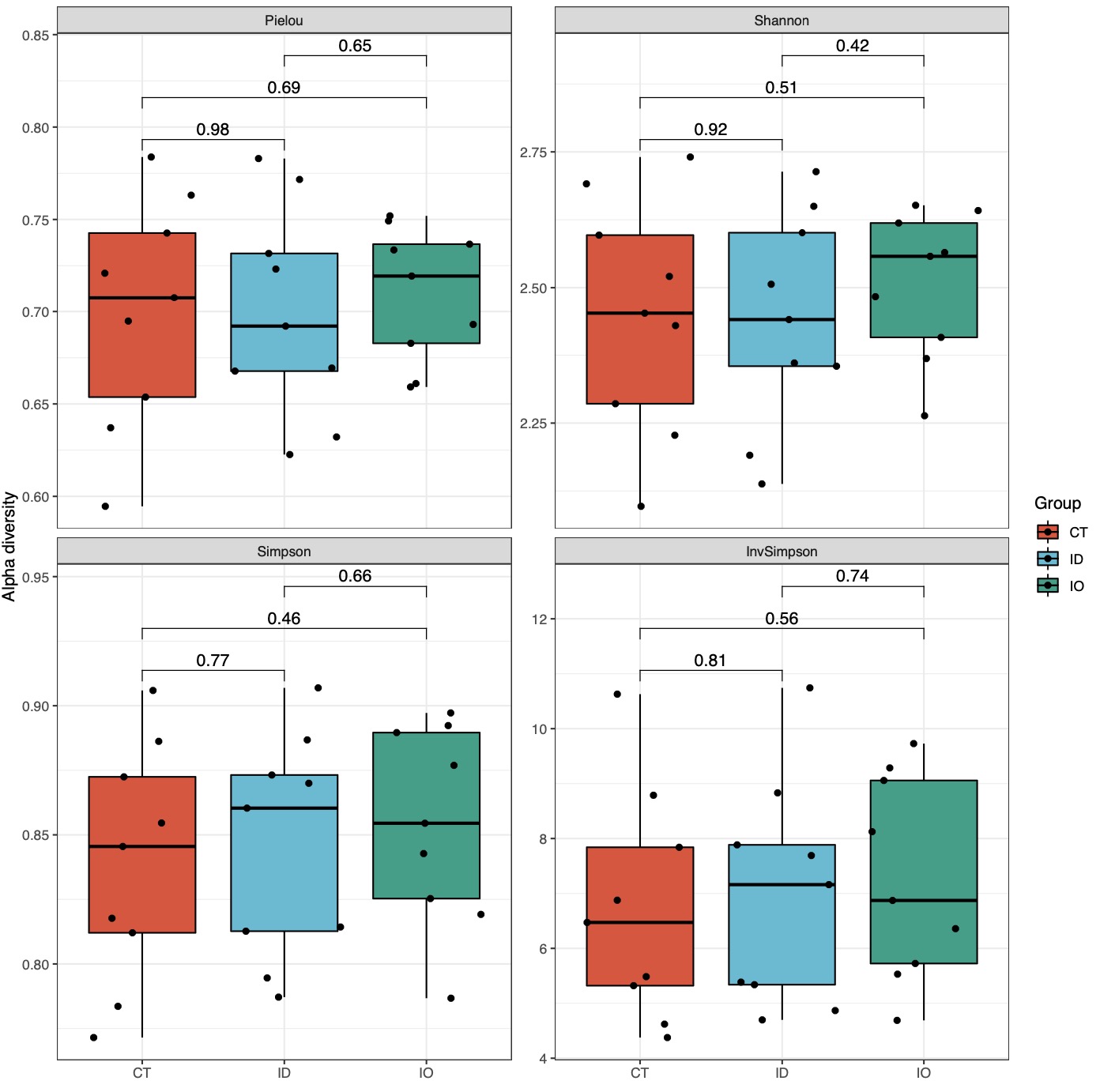
# 结果统计支持多组间onewayANOVA和事后多重检验Post-Hoc
# 需要设定method参数
alpha_re <- alpha_plot(dir = '16s_data/',design = 'mapping/mapping.txt',min_relative = 0.001,min_ratio = 0.7,
method = 'LSD')
alpha_re$plot$species$pic$Total
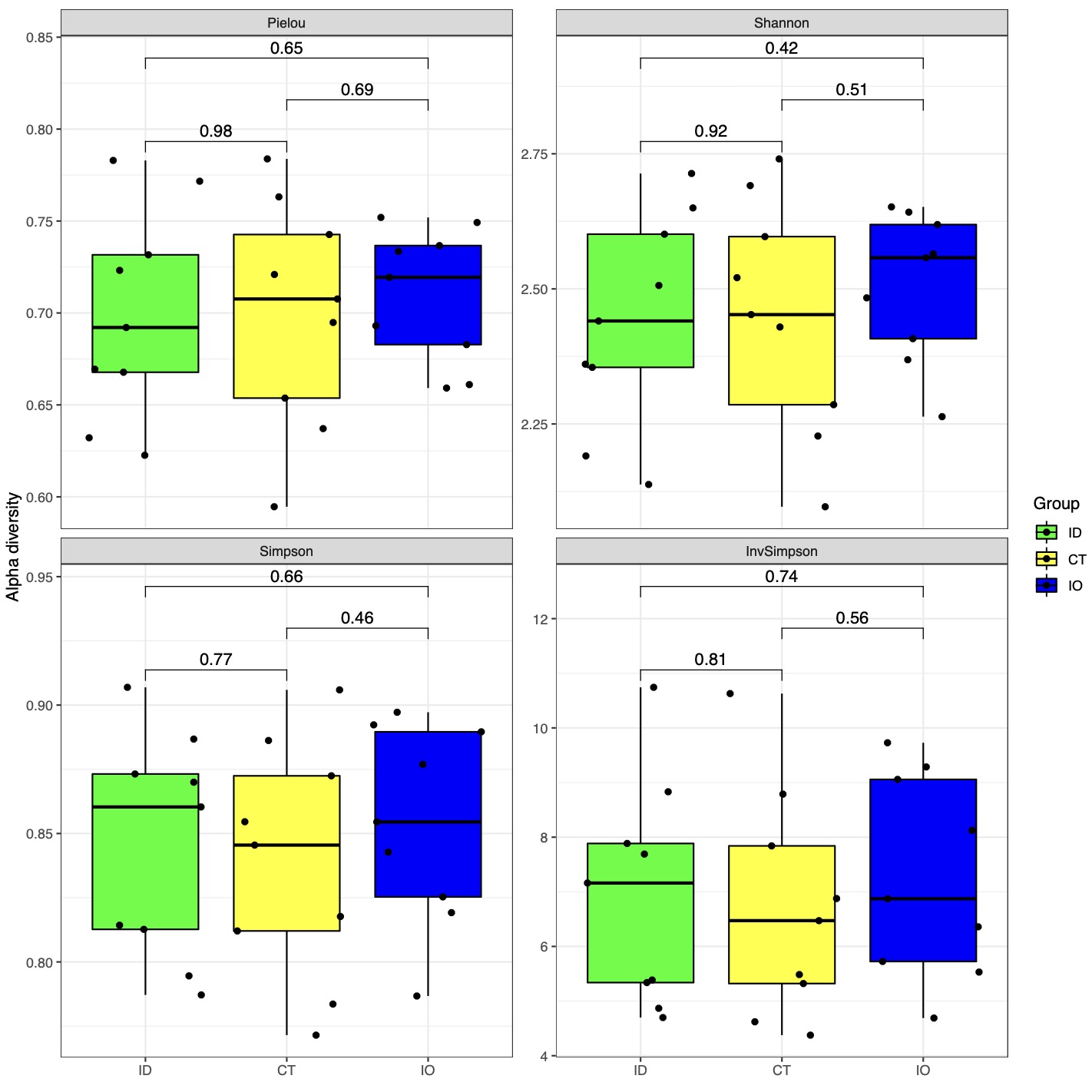
# 图形输出可以通过调整group_level参数和palette参数进行调整分组排序和颜色方案
group_order <- c('ID','CT','IO') ## 设定排序,名称需要绝对一致
cols <- c('green','yellow','blue') ## 设定颜色方案
alpha_re <- alpha_plot(dir = '16s_data/',design = 'mapping/mapping.txt',min_relative = 0.001,min_ratio = 0.7,
method = 'ttest',group_level = group_order,palette = cols)
alpha_re$plot$species$pic$Total
# alpha_plot支持ggplot2主题语法,可以使用mytheme参数进行进一步美化
library(ggplot2)
newtheme_slope=theme(axis.text.x =element_text(angle = 45, hjust = 1,size = 10)) # 例如调整分组名称的角度
alpha_re=alpha_plot(data = EMP$micro,design = EMP$mapping,min_relative = 0.001,min_ratio = 0.7,
method = 'ttest',mytheme = newtheme_slope)
alpha_re$plot$species$pic$Total